摘要:哈佛大學威斯生物激勵工程研究所的一項新進展克服了目前在空間轉錄組學方面的局限性,采用了一種名為“Light-Seq”的DNA納米技術驅動方法。Light-Seq允許研究人員用專屬于少數感興趣細胞的獨特DNA條形碼對全部RNA序列進行“地理標記”。
在顯微鏡下,研究人員經常觀察到不同類型的細胞在組織中以獨特的模式自我組織,或者有時一種罕見的細胞類型通過占據獨特的位置、顯示出不尋常的形狀或表達特定的生物標記分子而脫穎而出。為了確定他們觀察到的更深層次的意義,他們開發了一種方法,通過分析細胞中存在的基因來源的RNA分子來訪問細胞的基因表達模式(轉錄組),他們可以將其與細胞的形狀、空間位置和分子生物標記進行匹配。
然而,這些“空間轉錄組學”方法仍然只能捕獲細胞總RNA分子的一小部分,不能提供單細胞測序方法提供的深度和質量分析,單細胞測序方法是為了研究從組織或生物液體中分離出來的單個細胞的轉錄組而開發的通過下一代測序(NGS)技術。它們也不允許研究人員僅根據細胞在組織中的位置鎖定特定細胞,這將極大地促進研究分離的細胞群,或罕見的、難以分離的細胞,如具有獨特功能的罕見腦細胞,或入侵腫瘤的免疫細胞。此外,由于原始組織環境被破壞,許多空間轉錄組學和所有單細胞測序方法使研究人員無法重新訪問樣本進行后續分析,而且這些方法需要專門的儀器或試劑,因此成本很高。
哈佛大學威斯生物激勵工程研究所的一項新進展通過一種名為“Light- seq”的DNA納米技術驅動方法克服了這些限制。Light-Seq允許研究人員用專屬于少數感興趣細胞的獨特DNA條形碼對全部RNA序列進行“地理標記”。這些靶細胞在顯微鏡下通過一個快速有效的光交聯過程被選擇。

圖1 Light-Seq允許用專屬于少數細胞的獨特DNA條形碼對全部RNA序列進行標記(圖源:[1])
在一種新的DNA納米技術的幫助下,條形碼RNA序列被翻譯成連貫的DNA鏈,然后可以從組織樣本中收集并使用NGS進行識別。Light-Seq過程可以在相同的樣本中對不同的細胞群使用不同的條形碼進行重復,并保留原樣以供后續分析。它的性能可與單細胞測序方法相媲美,極大地拓寬了組織樣本調查的深度和范圍。
通訊作者之一、懷斯研究所核心成員Peng Yin博士說:“Light-Seq獨特的功能組合填補了未滿足的需求:對保存的組織中難以分離的細胞群或罕見細胞類型進行圖像信息、空間規定、深度測序分析的能力,與它們高度細化的基因表達狀態與空間、形態和潛在疾病相關特征的一一對應,因此,它有潛力快速推進生物醫學研究領域的生物發現過程。”

圖2 Light Seq支持對選定的細胞或組織區域原位進行選擇性條碼用于轉錄組測序(圖源:[1])
從原位條碼到異位測序
Light-Seq項目是由Yin在Wyss的小組中的Jocelyn (Josie) Kishi博士、Sinem Saka博士和Ninning Liu博士以及Constance Cepko在HMS的實驗室中的Emma West博士帶頭發起的。
在此之前,Kishi和Saka已經開發了SABER-FISH,作為一種空間轉錄組學方法,直接在完整的組織中(原位)對基因表達進行成像。"有了SABER-FISH,我們離捕獲細胞的完整基因表達程序仍有幾個數量級的距離,每個細胞有成千上萬的不同的RNA分子。RNA分子過于密集,無法用目前的成像技術完整地捕獲它們,"共同第一作者和共同通訊作者Kishi說。"Light-Seq通過將高分辨率的條形碼標記與通過NGS進行的全轉錄組測序結合起來解決了這個問題,使我們獲得了兩個世界的最好結果和額外的關鍵優勢。" 在進行這項研究時,Kishi是Yin團隊中的一名Wyss技術發展研究員,現在正與她的一些合著者一起追求Light-Seq的商業化道路。
“為了在完整組織樣本的定制選擇位置對細胞進行特定測序,我們開發了一種新的方法,將DNA條形碼光交聯到RNA分子的副本上,以及一種DNA納米技術驅動的程序,使它們及其附加的RNA序列可被NGS讀取。”共同第一作者Liu說,他是Yin團隊的博士后,之前曾與人合作開發了一個名為“Action-PAINT”的超分辨率成像方法的并行DNA條形碼平臺,該平臺也成為了Light-Seq的核心組件之一。
首先,DNA引物與細胞中的RNA分子“堿基對”,并被擴展以創建稱為互補DNA序列(cdna)的RNA序列副本。然后,含有超快光交聯子核苷酸的DNA條形碼鏈依次與細胞中的cdna進行堿基配對。當靶細胞在顯微鏡下通過一種類似模板的光學裝置被照亮時,這些細胞就會永久連接在一起,這種光學裝置將顯微鏡下的其他非靶細胞置于黑暗中,從而使它們免受光交聯反應的影響。在洗掉那些沒有永久連接的細胞中的條形碼DNA序列之后原位,這個過程可以用不同的條形碼和光線模式重復,以標記更多感興趣的區域。
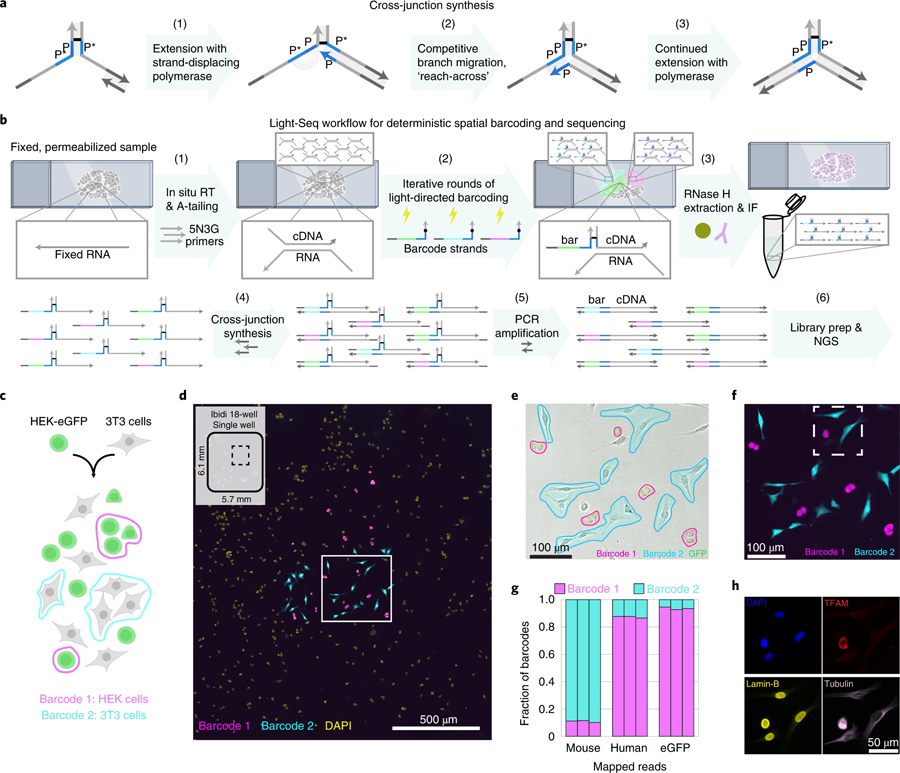
圖3 交聯合成和結合細胞混合物驗證的全原位協議。(圖源:[1])
“為了能夠將這種條形碼工作流程與NGS集成,我們設計了一種基于DNA納米技術的新的拼接反應。這項創新使我們能夠將我們的條形碼cdna轉換成連續的讀出序列。然后我們可以從樣本中提取含有條形碼的cDNA序列的完整集合,并用標準的NGS技術分析它們,最終,每個條形碼都將完整的轉錄組讀數追溯到組織樣本中預先選擇的細胞,這些細胞保持完整,以供后續分析。這為我們提供了一個獨特的機會,在測序驗證或進一步探索后重新訪問完全相同的細胞。
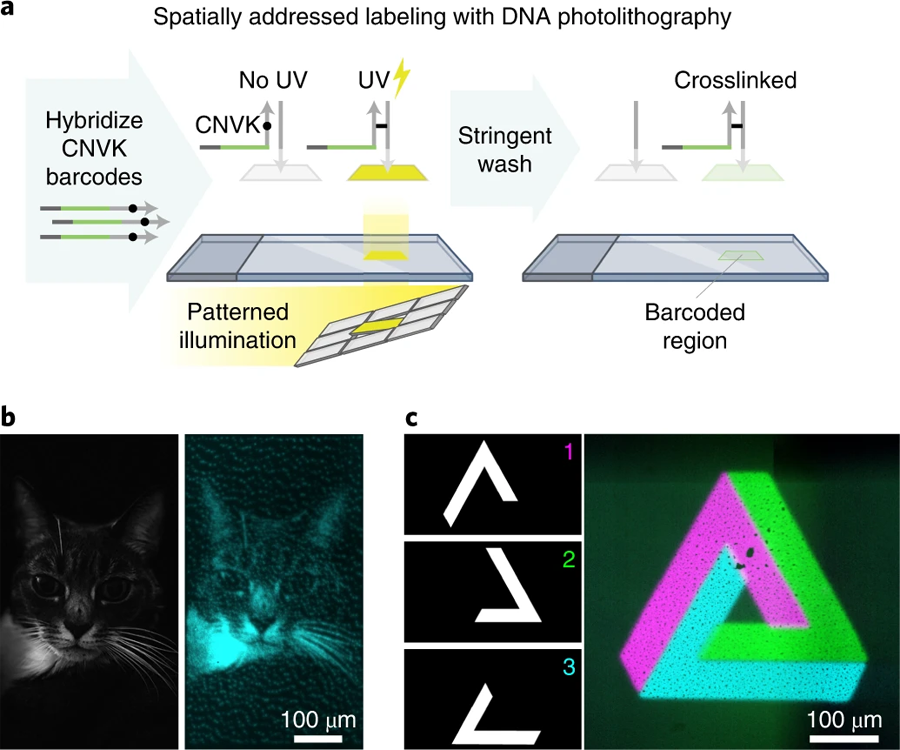
圖4 通過DNA光交聯進行空間定位標記(圖源:[1])
a 生物素標記的單鏈DNA寡核苷酸通過生物素-鏈霉親和素結合固定在玻璃表面。
觀察復雜組織和稀有細胞
隨著Light-Seq在培養細胞中的首次驗證,Yin的團隊想要將其應用到復雜的組織中,并與HMS的Constance Cepko博士團隊合作。Cepko是該研究的通訊作者之一,他研究了視網膜作為神經系統模型的發展。Kishi, Saka和Liu與Cepko團隊的West合作,將Light-Seq應用于小鼠視網膜的橫截面,并分析具有不同功能的三個主要層。研究人員達到了與單細胞測序方法相當的序列覆蓋范圍,并發現視網膜的三個主要層之間富集了數千個rna。他們還表明,在序列提取后,組織樣本保持完整,并可以進一步成像的蛋白質和其他生物分子。
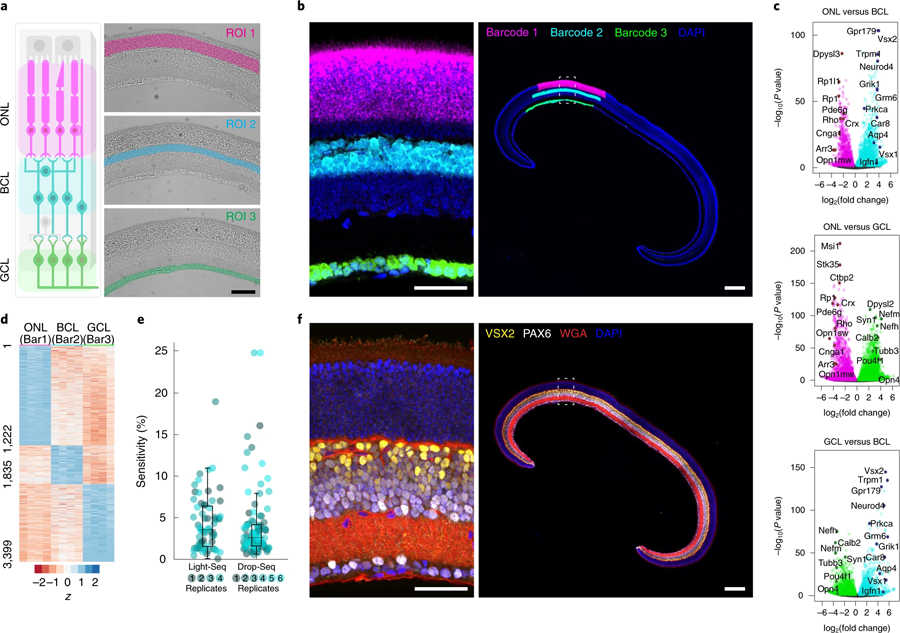
圖5 Light Seq技術應用于小鼠冷凍視網膜固定區域的三個主要視網膜層的空間條碼編碼。(圖源:[1])
West說:“將Light-Seq發揮到極致,我們能夠分離出一種非常罕見的細胞類型的完整轉錄組,這種細胞被稱為‘多巴胺能無分泌細胞’(dac),由于它與視網膜中其他細胞的復雜連接,很難分離,每個橫截面只提取4到8個獨立的條形碼細胞。”dac參與調節眼睛的晝夜節律,通過微調晝夜周期中不同光照的視覺感知。West補充說:“Light-Seq還檢測到在低水平下特異表達在dac中的rna,以及幾十種dac特異性的生物標志物rna,據我們所知,這為研究這種罕見的細胞類型打開了新的機會。”West在進行這項研究時是Cepko的研究生,然后是博士后,現在已經加入了Kishi的Light-Seq商業化努力。
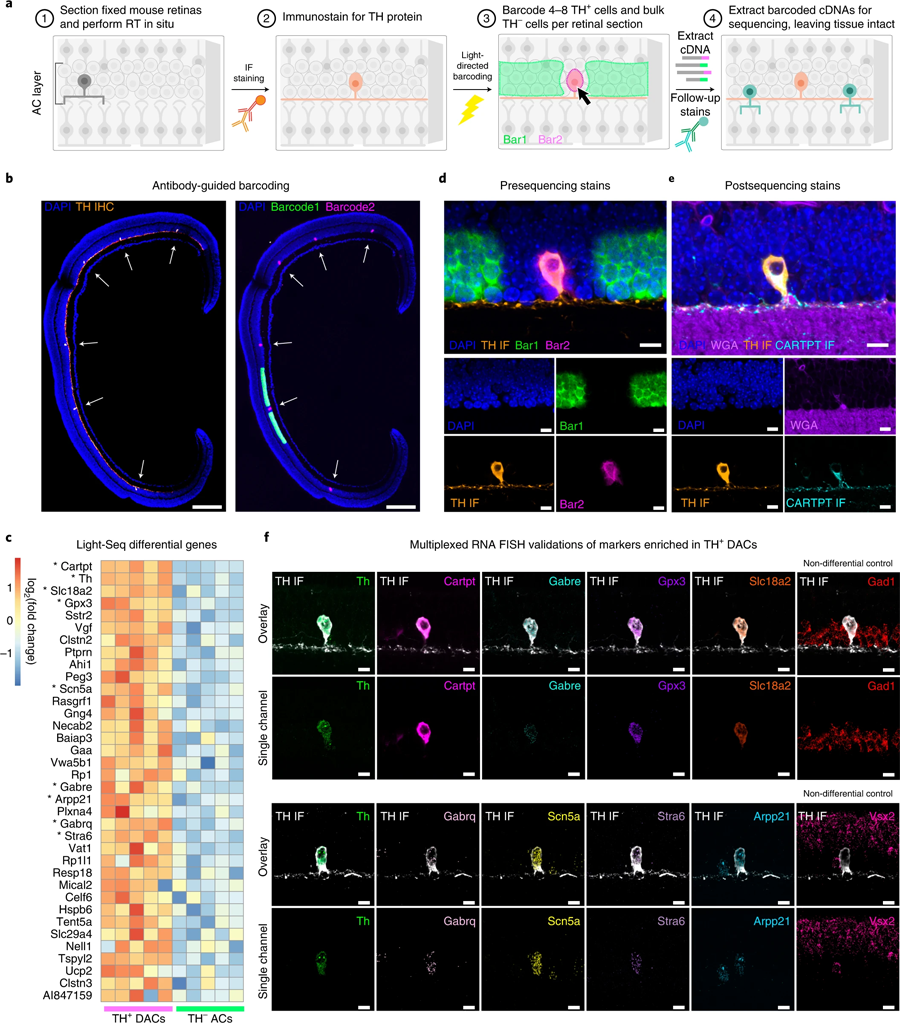
圖6 Light Seq用于稀有細胞轉錄組學(圖源:[1])
將空間轉錄組學領域向NGS開放,也增加了單個RNA物種水平的信息。“我們的測序數據清楚地表明,Light-Seq可以確定rna結構的自然變化。展望未來,我們對使用Light-Seq更好地了解免疫系統、疾病傳播細胞和不同治療策略(如基因和細胞治療)之間的相互作用非常感興趣。”Kishi說。
“由Peng Yin的團隊在Wyss研究所的分子機器人計劃中開發的Light-Seq技術再次表明,追求完全非傳統的方法和利用合成生物學可以導致一種具有巨大潛力的顛覆性技術,促進基礎研究和臨床醫學的發展。”
參考資料:
[1] Light-Seq: Light-directed in situ barcoding of biomolecules in fixed cells and tissues for spatially indexed sequencing
摘要:哈佛大學威斯生物激勵工程研究所的一項新進展克服了目前在空間轉錄組學方面的局限性,采用了一種名為“Light-Seq”的DNA納米技術驅動方法。Light-Seq允許研究人員用專屬于少數感興趣細胞的獨特DNA條形碼對全部RNA序列進行“地理標記”。
在顯微鏡下,研究人員經常觀察到不同類型的細胞在組織中以獨特的模式自我組織,或者有時一種罕見的細胞類型通過占據獨特的位置、顯示出不尋常的形狀或表達特定的生物標記分子而脫穎而出。為了確定他們觀察到的更深層次的意義,他們開發了一種方法,通過分析細胞中存在的基因來源的RNA分子來訪問細胞的基因表達模式(轉錄組),他們可以將其與細胞的形狀、空間位置和分子生物標記進行匹配。
然而,這些“空間轉錄組學”方法仍然只能捕獲細胞總RNA分子的一小部分,不能提供單細胞測序方法提供的深度和質量分析,單細胞測序方法是為了研究從組織或生物液體中分離出來的單個細胞的轉錄組而開發的通過下一代測序(NGS)技術。它們也不允許研究人員僅根據細胞在組織中的位置鎖定特定細胞,這將極大地促進研究分離的細胞群,或罕見的、難以分離的細胞,如具有獨特功能的罕見腦細胞,或入侵腫瘤的免疫細胞。此外,由于原始組織環境被破壞,許多空間轉錄組學和所有單細胞測序方法使研究人員無法重新訪問樣本進行后續分析,而且這些方法需要專門的儀器或試劑,因此成本很高。
哈佛大學威斯生物激勵工程研究所的一項新進展通過一種名為“Light- seq”的DNA納米技術驅動方法克服了這些限制。Light-Seq允許研究人員用專屬于少數感興趣細胞的獨特DNA條形碼對全部RNA序列進行“地理標記”。這些靶細胞在顯微鏡下通過一個快速有效的光交聯過程被選擇。

圖1 Light-Seq允許用專屬于少數細胞的獨特DNA條形碼對全部RNA序列進行標記(圖源:[1])
在一種新的DNA納米技術的幫助下,條形碼RNA序列被翻譯成連貫的DNA鏈,然后可以從組織樣本中收集并使用NGS進行識別。Light-Seq過程可以在相同的樣本中對不同的細胞群使用不同的條形碼進行重復,并保留原樣以供后續分析。它的性能可與單細胞測序方法相媲美,極大地拓寬了組織樣本調查的深度和范圍。
通訊作者之一、懷斯研究所核心成員Peng Yin博士說:“Light-Seq獨特的功能組合填補了未滿足的需求:對保存的組織中難以分離的細胞群或罕見細胞類型進行圖像信息、空間規定、深度測序分析的能力,與它們高度細化的基因表達狀態與空間、形態和潛在疾病相關特征的一一對應,因此,它有潛力快速推進生物醫學研究領域的生物發現過程。”

圖2 Light Seq支持對選定的細胞或組織區域原位進行選擇性條碼用于轉錄組測序(圖源:[1])
從原位條碼到異位測序
Light-Seq項目是由Yin在Wyss的小組中的Jocelyn (Josie) Kishi博士、Sinem Saka博士和Ninning Liu博士以及Constance Cepko在HMS的實驗室中的Emma West博士帶頭發起的。
在此之前,Kishi和Saka已經開發了SABER-FISH,作為一種空間轉錄組學方法,直接在完整的組織中(原位)對基因表達進行成像。"有了SABER-FISH,我們離捕獲細胞的完整基因表達程序仍有幾個數量級的距離,每個細胞有成千上萬的不同的RNA分子。RNA分子過于密集,無法用目前的成像技術完整地捕獲它們,"共同第一作者和共同通訊作者Kishi說。"Light-Seq通過將高分辨率的條形碼標記與通過NGS進行的全轉錄組測序結合起來解決了這個問題,使我們獲得了兩個世界的最好結果和額外的關鍵優勢。" 在進行這項研究時,Kishi是Yin團隊中的一名Wyss技術發展研究員,現在正與她的一些合著者一起追求Light-Seq的商業化道路。
“為了在完整組織樣本的定制選擇位置對細胞進行特定測序,我們開發了一種新的方法,將DNA條形碼光交聯到RNA分子的副本上,以及一種DNA納米技術驅動的程序,使它們及其附加的RNA序列可被NGS讀取。”共同第一作者Liu說,他是Yin團隊的博士后,之前曾與人合作開發了一個名為“Action-PAINT”的超分辨率成像方法的并行DNA條形碼平臺,該平臺也成為了Light-Seq的核心組件之一。
首先,DNA引物與細胞中的RNA分子“堿基對”,并被擴展以創建稱為互補DNA序列(cdna)的RNA序列副本。然后,含有超快光交聯子核苷酸的DNA條形碼鏈依次與細胞中的cdna進行堿基配對。當靶細胞在顯微鏡下通過一種類似模板的光學裝置被照亮時,這些細胞就會永久連接在一起,這種光學裝置將顯微鏡下的其他非靶細胞置于黑暗中,從而使它們免受光交聯反應的影響。在洗掉那些沒有永久連接的細胞中的條形碼DNA序列之后原位,這個過程可以用不同的條形碼和光線模式重復,以標記更多感興趣的區域。

圖3 交聯合成和結合細胞混合物驗證的全原位協議。(圖源:[1])
“為了能夠將這種條形碼工作流程與NGS集成,我們設計了一種基于DNA納米技術的新的拼接反應。這項創新使我們能夠將我們的條形碼cdna轉換成連續的讀出序列。然后我們可以從樣本中提取含有條形碼的cDNA序列的完整集合,并用標準的NGS技術分析它們,最終,每個條形碼都將完整的轉錄組讀數追溯到組織樣本中預先選擇的細胞,這些細胞保持完整,以供后續分析。這為我們提供了一個獨特的機會,在測序驗證或進一步探索后重新訪問完全相同的細胞。

圖4 通過DNA光交聯進行空間定位標記(圖源:[1])
a 生物素標記的單鏈DNA寡核苷酸通過生物素-鏈霉親和素結合固定在玻璃表面。
觀察復雜組織和稀有細胞
隨著Light-Seq在培養細胞中的首次驗證,Yin的團隊想要將其應用到復雜的組織中,并與HMS的Constance Cepko博士團隊合作。Cepko是該研究的通訊作者之一,他研究了視網膜作為神經系統模型的發展。Kishi, Saka和Liu與Cepko團隊的West合作,將Light-Seq應用于小鼠視網膜的橫截面,并分析具有不同功能的三個主要層。研究人員達到了與單細胞測序方法相當的序列覆蓋范圍,并發現視網膜的三個主要層之間富集了數千個rna。他們還表明,在序列提取后,組織樣本保持完整,并可以進一步成像的蛋白質和其他生物分子。

圖5 Light Seq技術應用于小鼠冷凍視網膜固定區域的三個主要視網膜層的空間條碼編碼。(圖源:[1])
West說:“將Light-Seq發揮到極致,我們能夠分離出一種非常罕見的細胞類型的完整轉錄組,這種細胞被稱為‘多巴胺能無分泌細胞’(dac),由于它與視網膜中其他細胞的復雜連接,很難分離,每個橫截面只提取4到8個獨立的條形碼細胞。”dac參與調節眼睛的晝夜節律,通過微調晝夜周期中不同光照的視覺感知。West補充說:“Light-Seq還檢測到在低水平下特異表達在dac中的rna,以及幾十種dac特異性的生物標志物rna,據我們所知,這為研究這種罕見的細胞類型打開了新的機會。”West在進行這項研究時是Cepko的研究生,然后是博士后,現在已經加入了Kishi的Light-Seq商業化努力。

圖6 Light Seq用于稀有細胞轉錄組學(圖源:[1])
將空間轉錄組學領域向NGS開放,也增加了單個RNA物種水平的信息。“我們的測序數據清楚地表明,Light-Seq可以確定rna結構的自然變化。展望未來,我們對使用Light-Seq更好地了解免疫系統、疾病傳播細胞和不同治療策略(如基因和細胞治療)之間的相互作用非常感興趣。”Kishi說。
“由Peng Yin的團隊在Wyss研究所的分子機器人計劃中開發的Light-Seq技術再次表明,追求完全非傳統的方法和利用合成生物學可以導致一種具有巨大潛力的顛覆性技術,促進基礎研究和臨床醫學的發展。”
參考資料:
[1] Light-Seq: Light-directed in situ barcoding of biomolecules in fixed cells and tissues for spatially indexed sequencing



