摘要:研究人員采用了一種名為堿基特異性原位測序(BaSISS)的新方法,能夠定量繪制腫瘤克隆的遺傳組成及其空間結(jié)構(gòu)。
癌癥的基因組測序顯示,不同的亞克隆往往鑲嵌在同一腫瘤中。盡管這些是根據(jù)體細(xì)胞進(jìn)化的原理產(chǎn)生的,但確切的空間生長模式和背后的潛在機(jī)制還不大清楚。
近日,一個(gè)國際研究團(tuán)隊(duì)利用空間基因組學(xué)技術(shù)繪制了兩名患者乳腺腫瘤中的不同克隆。這項(xiàng)研究成果于11月9日發(fā)表在《Nature》雜志上,為腫瘤如何進(jìn)展以及如何與周圍環(huán)境相互作用提供了新見解。
在這項(xiàng)研究中,研究人員采用了一種名為堿基特異性原位測序(BaSISS)的新方法。通過這種方法,他們能夠定量繪制腫瘤克隆的遺傳組成及其空間結(jié)構(gòu)。

圖1 研究人員采用了一種名為堿基特異性原位測序(BaSISS)的新方法(圖源:[1])
研究團(tuán)隊(duì)采用這種方法來分析兩名多灶性乳腺癌患者的八個(gè)組織樣本,這些樣本來自癌癥進(jìn)展的不同階段。他們發(fā)現(xiàn),不同克隆在樣本中顯示出一定的空間結(jié)構(gòu),具有特定的基因表達(dá)模式以及微環(huán)境和微解剖生態(tài)位。
共同通訊作者、瑞典斯德哥爾摩大學(xué)生命科學(xué)實(shí)驗(yàn)室的Mats Nilsson教授表示:“通過這項(xiàng)創(chuàng)新技術(shù),我們能夠準(zhǔn)確地重建這些克隆的擴(kuò)散過程。我們在研究后得出的一個(gè)重要結(jié)論是,癌細(xì)胞存活和擴(kuò)散的原因不僅僅在于遺傳變化,可能也與它們所在的位置有關(guān)。這又增添了一層復(fù)雜性,同時(shí)也為疾病治療帶來了潛在新方法。”
Mats Nilsson教授是原位測序領(lǐng)域的先鋒人物,其團(tuán)隊(duì)開發(fā)出多種原位測序方法,這些方法已通過Cartana公司商業(yè)化。2020年,Cartana公司被10x Genomics收購,而Mats Nilsson成為10x Genomics的科學(xué)顧問。
BaSISS的流程是將新鮮冷凍的組織塊進(jìn)行冷凍切片,然后進(jìn)行空間克隆定位和表型分析。在利用批量細(xì)胞測序發(fā)現(xiàn)新發(fā)突變和腫瘤亞克隆后,他們設(shè)計(jì)了特定的熒光分子探針來檢測轉(zhuǎn)錄本在切片上的哪個(gè)位置表達(dá)。之后通過計(jì)算機(jī)方法生成克隆圖譜。
Nilsson及其同事將這種方法應(yīng)用在兩名患者的八個(gè)組織塊上,其中一名患者患有ER陽性而HER陰性的原發(fā)性浸潤性乳腺癌,另一名患者患有三陰性浸潤性乳腺癌。這些組織樣本覆蓋了癌癥進(jìn)展的各個(gè)階段,從導(dǎo)管原位癌一直到浸潤性癌癥和淋巴結(jié)轉(zhuǎn)移。

圖2 BaSISS繪制癌癥克隆的工作流程(圖源:[1])
分析結(jié)果顯示,亞克隆生長呈現(xiàn)出復(fù)雜的模式。例如,研究人員發(fā)現(xiàn),亞克隆傾向于形成與其組織學(xué)進(jìn)展?fàn)顟B(tài)相關(guān)的空間模式。然而,每個(gè)亞克隆之間又存在數(shù)百個(gè)突變的區(qū)別,這些突變同時(shí)存在于原位癌和浸潤性組織學(xué)狀態(tài)中。這表明組織學(xué)和遺傳病進(jìn)展之間也存在脫節(jié)。
研究人員還指出,表型或組織學(xué)狀態(tài)與細(xì)胞的遺傳狀態(tài)之間存在關(guān)聯(lián)。例如,與野生型PTEN克隆相比,攜帶PTEN突變的克隆顯示出更高密度的 Ki-67 IHC細(xì)胞核染色。然而,對于任何克隆,無論是導(dǎo)管原位癌還是浸潤性狀態(tài),Ki-67評分都是相似的,表明盡管Ki-67上調(diào)的發(fā)生時(shí)間與PTEN突變時(shí)間相似,但它發(fā)生在腫瘤具有侵襲性之前。
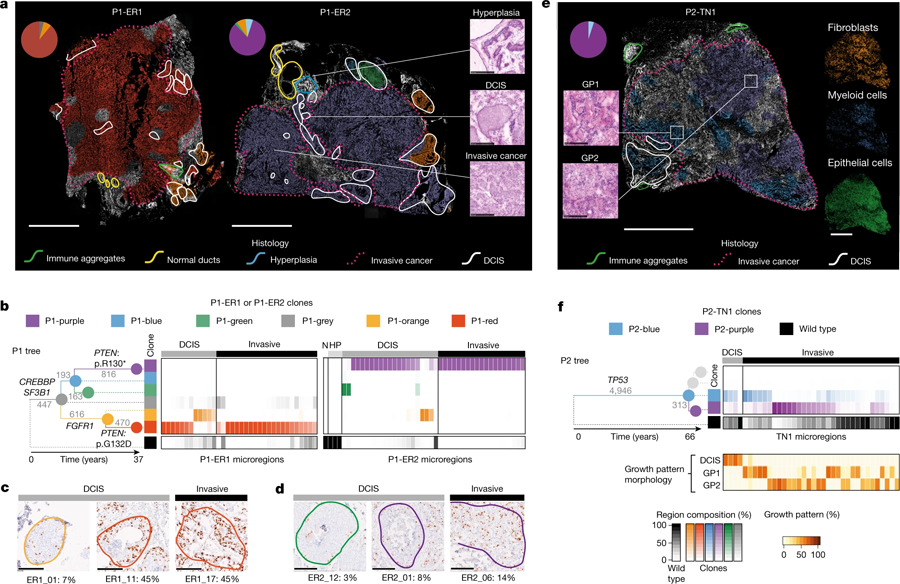
圖3 由三位原發(fā)性乳腺癌患者的組織學(xué)檢測繪制的遺傳克隆圖(圖源:[1])
德國癌癥研究中心的共同資深作者M(jìn)oritz Gerstung稱:“這項(xiàng)技術(shù)令人興奮的地方在于,我們第一次看到環(huán)境是如何影響癌癥進(jìn)化的。我們能夠看到哪些癌癥克隆變得更具侵襲性,而哪些沒有,這將幫助我們更好地了解腫瘤生長的關(guān)鍵步驟,以及我們該如何減輕或預(yù)防疾病。”
參考資料:
[1] Lomakin, A., Svedlund, J., Strell, C. et al. Spatial genomics maps the structure, nature and evolution of cancer clones. Nature (2022). https://doi.org/10.1038/s41586-022-05425-2
摘要:研究人員采用了一種名為堿基特異性原位測序(BaSISS)的新方法,能夠定量繪制腫瘤克隆的遺傳組成及其空間結(jié)構(gòu)。
癌癥的基因組測序顯示,不同的亞克隆往往鑲嵌在同一腫瘤中。盡管這些是根據(jù)體細(xì)胞進(jìn)化的原理產(chǎn)生的,但確切的空間生長模式和背后的潛在機(jī)制還不大清楚。
近日,一個(gè)國際研究團(tuán)隊(duì)利用空間基因組學(xué)技術(shù)繪制了兩名患者乳腺腫瘤中的不同克隆。這項(xiàng)研究成果于11月9日發(fā)表在《Nature》雜志上,為腫瘤如何進(jìn)展以及如何與周圍環(huán)境相互作用提供了新見解。
在這項(xiàng)研究中,研究人員采用了一種名為堿基特異性原位測序(BaSISS)的新方法。通過這種方法,他們能夠定量繪制腫瘤克隆的遺傳組成及其空間結(jié)構(gòu)。

圖1 研究人員采用了一種名為堿基特異性原位測序(BaSISS)的新方法(圖源:[1])
研究團(tuán)隊(duì)采用這種方法來分析兩名多灶性乳腺癌患者的八個(gè)組織樣本,這些樣本來自癌癥進(jìn)展的不同階段。他們發(fā)現(xiàn),不同克隆在樣本中顯示出一定的空間結(jié)構(gòu),具有特定的基因表達(dá)模式以及微環(huán)境和微解剖生態(tài)位。
共同通訊作者、瑞典斯德哥爾摩大學(xué)生命科學(xué)實(shí)驗(yàn)室的Mats Nilsson教授表示:“通過這項(xiàng)創(chuàng)新技術(shù),我們能夠準(zhǔn)確地重建這些克隆的擴(kuò)散過程。我們在研究后得出的一個(gè)重要結(jié)論是,癌細(xì)胞存活和擴(kuò)散的原因不僅僅在于遺傳變化,可能也與它們所在的位置有關(guān)。這又增添了一層復(fù)雜性,同時(shí)也為疾病治療帶來了潛在新方法。”
Mats Nilsson教授是原位測序領(lǐng)域的先鋒人物,其團(tuán)隊(duì)開發(fā)出多種原位測序方法,這些方法已通過Cartana公司商業(yè)化。2020年,Cartana公司被10x Genomics收購,而Mats Nilsson成為10x Genomics的科學(xué)顧問。
BaSISS的流程是將新鮮冷凍的組織塊進(jìn)行冷凍切片,然后進(jìn)行空間克隆定位和表型分析。在利用批量細(xì)胞測序發(fā)現(xiàn)新發(fā)突變和腫瘤亞克隆后,他們設(shè)計(jì)了特定的熒光分子探針來檢測轉(zhuǎn)錄本在切片上的哪個(gè)位置表達(dá)。之后通過計(jì)算機(jī)方法生成克隆圖譜。
Nilsson及其同事將這種方法應(yīng)用在兩名患者的八個(gè)組織塊上,其中一名患者患有ER陽性而HER陰性的原發(fā)性浸潤性乳腺癌,另一名患者患有三陰性浸潤性乳腺癌。這些組織樣本覆蓋了癌癥進(jìn)展的各個(gè)階段,從導(dǎo)管原位癌一直到浸潤性癌癥和淋巴結(jié)轉(zhuǎn)移。

圖2 BaSISS繪制癌癥克隆的工作流程(圖源:[1])
分析結(jié)果顯示,亞克隆生長呈現(xiàn)出復(fù)雜的模式。例如,研究人員發(fā)現(xiàn),亞克隆傾向于形成與其組織學(xué)進(jìn)展?fàn)顟B(tài)相關(guān)的空間模式。然而,每個(gè)亞克隆之間又存在數(shù)百個(gè)突變的區(qū)別,這些突變同時(shí)存在于原位癌和浸潤性組織學(xué)狀態(tài)中。這表明組織學(xué)和遺傳病進(jìn)展之間也存在脫節(jié)。
研究人員還指出,表型或組織學(xué)狀態(tài)與細(xì)胞的遺傳狀態(tài)之間存在關(guān)聯(lián)。例如,與野生型PTEN克隆相比,攜帶PTEN突變的克隆顯示出更高密度的 Ki-67 IHC細(xì)胞核染色。然而,對于任何克隆,無論是導(dǎo)管原位癌還是浸潤性狀態(tài),Ki-67評分都是相似的,表明盡管Ki-67上調(diào)的發(fā)生時(shí)間與PTEN突變時(shí)間相似,但它發(fā)生在腫瘤具有侵襲性之前。

圖3 由三位原發(fā)性乳腺癌患者的組織學(xué)檢測繪制的遺傳克隆圖(圖源:[1])
德國癌癥研究中心的共同資深作者M(jìn)oritz Gerstung稱:“這項(xiàng)技術(shù)令人興奮的地方在于,我們第一次看到環(huán)境是如何影響癌癥進(jìn)化的。我們能夠看到哪些癌癥克隆變得更具侵襲性,而哪些沒有,這將幫助我們更好地了解腫瘤生長的關(guān)鍵步驟,以及我們該如何減輕或預(yù)防疾病。”
參考資料:
[1] Lomakin, A., Svedlund, J., Strell, C. et al. Spatial genomics maps the structure, nature and evolution of cancer clones. Nature (2022). https://doi.org/10.1038/s41586-022-05425-2



