摘要:每一個CRISPR反應的核心,都是通過引導RNA到DNA上的目標位點的Cas蛋白質的強分子連接。
每一個CRISPR反應的核心,無論是自然發生在細菌中,還是通過CRIPSR-Cas基因編輯技術加以利用,都是通過引導RNA到達DNA上的目標位點,使Cas蛋白質形成一個強大的分子鍵。
霍華德·休斯醫學研究所研究員Michelle Wang說:“在穩定的邊界和在正確的時間脫離之間有一個平衡。”“我們真正想要的是調節結合力的能力。這給了我們微調基因編輯潛力的可能性。”
Wang實驗室的博士生、該論文的主要作者Porter Hall認為,Cas蛋白的結合不能太短暫。如果它不能穩定地結合DNA的目標區域,精確的基因編輯可能不會有效,可能會導致脫靶效應。“但如果蛋白質永遠停留在那里,那么基因編輯過程就無法完成,”Hall說。

圖1 RNA聚合酶如何可以不進行切割就識別DNA序列就去除一個結合的dCas 圖源:[1])
通過對Cas與DNA結合的精確分子水平機制的研究,Wang和他的同事們首次解釋了運動蛋白(RNA聚合酶)如何去除一個結合的dCas(一種經過工程改造的Cas),可以不進行切割就識別DNA序列。
這一見解揭示了如何調整Cas去除,有助于未來的CRISPR應用。
研究人員寫道:“要充分發揮CRISPR技術的潛力,對Cas結合穩定性的深入機制理解至關重要。”“這項工作強調了R-loop在dCas結合穩定性中的重要性,并為CRISPR技術的廣泛應用提供了有價值的機制見解。”
Wang實驗室研究馬達蛋白如何沿著DNA鏈移動,執行重要的生物過程。
Wang說,馬達蛋白RNA聚合酶在執行基因表達功能時,會對“路障”施加壓力,將DNA復制到RNA。在本研究中,這個路障就是內切酶缺陷Cas (dCas)。
此前,研究人員使用納米光子鑷子機械地分離了兩條DNA鏈,繪制出結合的dCas蛋白質在DNA上的位置。他們稱之為DNA unzipping mapper。
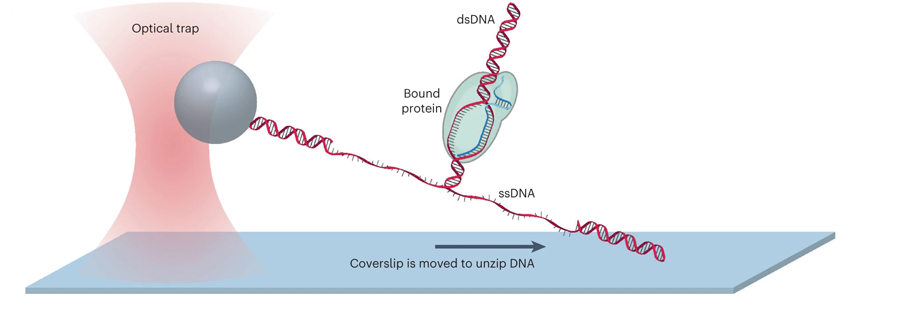
圖2 使用DNA解壓縮映射器繪制dCas與DNA相互作用的高分辨率圖 (圖源:[1])
先前的研究表明,馬達蛋白只可能從一側(極性)去除dCas。使用當前研究的DNA unzipping mapper,康奈爾大學的研究人員發現了其中的原因:因為RNA聚合酶只能從一側折疊結合dCas的引導RNA和目標DNA之間形成的環(稱為 "R-loop"),即距離PAM(protospacer adjacent motif)遠的一側(或遠處),PAM是一個2-6個堿基對長的短DNA序列,緊隨裂解目標DNA區域。
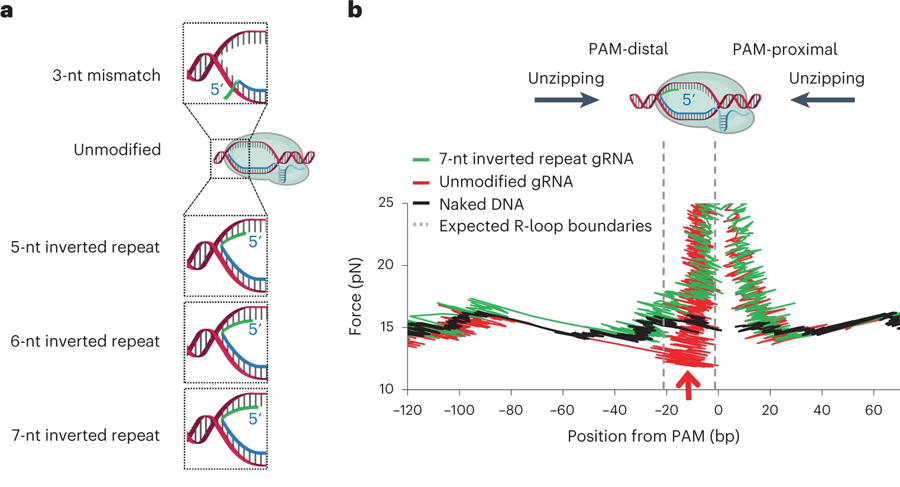
圖3 結合dCas復合物的轉錄讀取的定量測定(圖源:[1])
研究人員描述了該機制是如何工作的,他們也展示了如何通過修改引導RNA來調整dCas R-loop的穩定性。
“我們希望Cas蛋白質如何工作的基礎知識最終能導致更有效的基因編輯和更廣泛的CRISPR技術應用,”Wang說。
參考資料:
[1] Polarity of the CRISPR Roadblock to Transcription
摘要:每一個CRISPR反應的核心,都是通過引導RNA到DNA上的目標位點的Cas蛋白質的強分子連接。
每一個CRISPR反應的核心,無論是自然發生在細菌中,還是通過CRIPSR-Cas基因編輯技術加以利用,都是通過引導RNA到達DNA上的目標位點,使Cas蛋白質形成一個強大的分子鍵。
霍華德·休斯醫學研究所研究員Michelle Wang說:“在穩定的邊界和在正確的時間脫離之間有一個平衡。”“我們真正想要的是調節結合力的能力。這給了我們微調基因編輯潛力的可能性。”
Wang實驗室的博士生、該論文的主要作者Porter Hall認為,Cas蛋白的結合不能太短暫。如果它不能穩定地結合DNA的目標區域,精確的基因編輯可能不會有效,可能會導致脫靶效應。“但如果蛋白質永遠停留在那里,那么基因編輯過程就無法完成,”Hall說。

圖1 RNA聚合酶如何可以不進行切割就識別DNA序列就去除一個結合的dCas 圖源:[1])
通過對Cas與DNA結合的精確分子水平機制的研究,Wang和他的同事們首次解釋了運動蛋白(RNA聚合酶)如何去除一個結合的dCas(一種經過工程改造的Cas),可以不進行切割就識別DNA序列。
這一見解揭示了如何調整Cas去除,有助于未來的CRISPR應用。
研究人員寫道:“要充分發揮CRISPR技術的潛力,對Cas結合穩定性的深入機制理解至關重要。”“這項工作強調了R-loop在dCas結合穩定性中的重要性,并為CRISPR技術的廣泛應用提供了有價值的機制見解。”
Wang實驗室研究馬達蛋白如何沿著DNA鏈移動,執行重要的生物過程。
Wang說,馬達蛋白RNA聚合酶在執行基因表達功能時,會對“路障”施加壓力,將DNA復制到RNA。在本研究中,這個路障就是內切酶缺陷Cas (dCas)。
此前,研究人員使用納米光子鑷子機械地分離了兩條DNA鏈,繪制出結合的dCas蛋白質在DNA上的位置。他們稱之為DNA unzipping mapper。

圖2 使用DNA解壓縮映射器繪制dCas與DNA相互作用的高分辨率圖 (圖源:[1])
先前的研究表明,馬達蛋白只可能從一側(極性)去除dCas。使用當前研究的DNA unzipping mapper,康奈爾大學的研究人員發現了其中的原因:因為RNA聚合酶只能從一側折疊結合dCas的引導RNA和目標DNA之間形成的環(稱為 "R-loop"),即距離PAM(protospacer adjacent motif)遠的一側(或遠處),PAM是一個2-6個堿基對長的短DNA序列,緊隨裂解目標DNA區域。

圖3 結合dCas復合物的轉錄讀取的定量測定(圖源:[1])
研究人員描述了該機制是如何工作的,他們也展示了如何通過修改引導RNA來調整dCas R-loop的穩定性。
“我們希望Cas蛋白質如何工作的基礎知識最終能導致更有效的基因編輯和更廣泛的CRISPR技術應用,”Wang說。
參考資料:
[1] Polarity of the CRISPR Roadblock to Transcription



